 属鱼类一新种(鲤形目, 鲤科)
属鱼类一新种(鲤形目, 鲤科)
 属鱼类一新种(鲤形目, 鲤科)
属鱼类一新种(鲤形目, 鲤科)
2. 上海海洋大学 环境DNA技术与水生态健康评估工程中心, 上海 201306;
3. 浙江省森林资源监测中心, 浙江 杭州 310020;
4. 浙江省林业勘测规划设计有限公司, 浙江 杭州 310020
 属(Zacco)鱼类一新种——苕溪
属(Zacco)鱼类一新种——苕溪 (Zacco tiaoxiensis sp. nov.)。经形态比较, 该新种与广泛分布于浙江各水系的棘颊
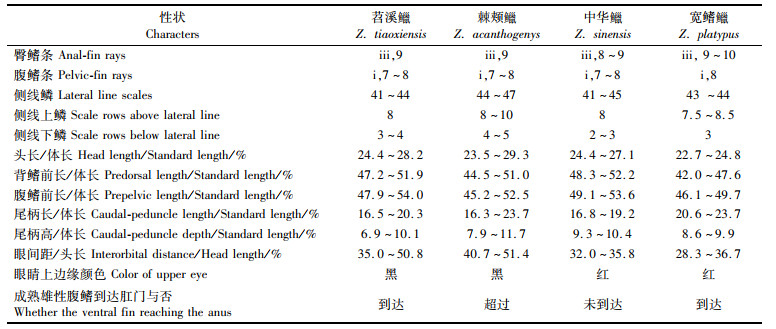
(Zacco tiaoxiensis sp. nov.)。经形态比较, 该新种与广泛分布于浙江各水系的棘颊 (Z. acanthogenys)形态较相似, 但与之有着较明显的形态差异: 侧线鳞41~44(vs. 44~47), 侧线上鳞8(vs. 8~10), 侧线下鳞3~4(vs. 4~5), 围尾柄鳞12~15(vs. 14~17), 性成熟的雄性个体腹鳍到达(vs. 超过)肛门。新种与异域分布的中华
(Z. acanthogenys)形态较相似, 但与之有着较明显的形态差异: 侧线鳞41~44(vs. 44~47), 侧线上鳞8(vs. 8~10), 侧线下鳞3~4(vs. 4~5), 围尾柄鳞12~15(vs. 14~17), 性成熟的雄性个体腹鳍到达(vs. 超过)肛门。新种与异域分布的中华 (Z.sinensis)和宽鳍
(Z.sinensis)和宽鳍 (Z.platypus)的形态区别主要在侧线下鳞数目、头长与体长的比例等可比性状以及眼睛上缘颜色等。基于线粒体细胞色素b基因的遗传分析也支持苕溪
(Z.platypus)的形态区别主要在侧线下鳞数目、头长与体长的比例等可比性状以及眼睛上缘颜色等。基于线粒体细胞色素b基因的遗传分析也支持苕溪 为一独立物种。
为一独立物种。 属 苕溪
属 苕溪 新种 细胞色素b 遗传距离 系统发育
新种 细胞色素b 遗传距离 系统发育 马口鱼类(Opsariichthine)是鲤形目(Cypriniformes)鲤科(Cyprinidae)中独特而复杂的一个类群,过去被归于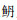















BOULENGER[12]曾根据采自浙江宁波的标本描述了马口鱼属一新种,命名为棘颊马口鱼(O. acanthogenys),陈宜瑜[3]将其视为宽鳍










在检视2021年6—9月采自浙江苕溪水系的



共采集到浙江苕溪水系

|
表 1 遗传分析所用样本及来源 Tab.1 Samples and sources for genetic analysis |
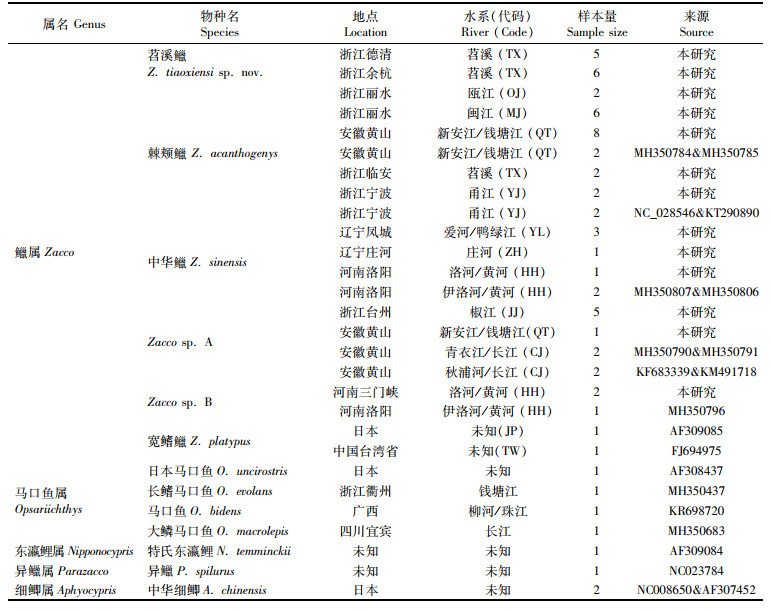
形态测量工作由同一人从鱼体的左侧进行,工具为数显游标卡尺,数据精确到0.1 mm,使用SPSS 25.0处理数据和计算平均值。形态计数和测量标准参考陈宜瑜[1]、朱兰等[15]、陈义雄等[16]。侧线上鳞的计数从背鳍起点前的侧面第1个鳞片开始,侧线下鳞为至腹鳍起点前的行数。对与新种形态最为相似的棘颊
采用上海生工生物工程股份有限公司的动物基因组DNA提取试剂盒提取基因组DNA,详细步骤参考说明书。Cytb基因的扩增及测序引物为正向引物L14724(5'-GACTTGAAAAACCACCGTTG-3')和反向引物H15915(5'-CTCCGATCTCCGGATTACAAGAC-3')[17]。RCR反应条件:95 ℃预变性3 min;94 ℃变性30 s;54 ℃退火45 s;72 ℃延伸60 s;35个循环;72 ℃延伸5 min;4 ℃保温。PCR产物用1%琼脂糖电泳检测样品扩增情况,送至上海生工生物工程股份有限公司完成双向测序。
1.3.2 数据分析经双向测序后,将得到的正反向两段序列使用DNAstar的SeqMan[18]软件进行拼接,并根据序列原始峰图进行手工校正。总共获得Cytb基因全序列44条,使用Clustal X[19]软件对拼接好的序列进行比对。
为了解新种在马口鱼类群中的系统发育位置,首先从GenBank下载21条序列,包括
苕溪,新种 Zacco tiaoxiensis sp. nov.(图版Ⅰ), 正模标本SHOU2021011012雄,体长96.9 mm;2021年11月采自浙江省杭州市余杭区留下街道小和山(苕溪水系, 120°00'31″E,30°12'46″N)。

|
1. 正模标本,SHOU2021011012,雄性,体长96.9 mm;2. 副模标本,SHOU2021011011,雌性,体长72.5 mm;3. 生活时雄性;4. 生活时雌性。
1. Holotype, preserved male specimen, SHOU2021011012, 96.9 mm standard length; 2. Paratype, preserved female specimen, SHOU2021011011, 72.5 mm standard length; 3. Alive male; 4. Alive female.
图版 新种苕溪 |
副模标本9尾: SHOU2021060252、SHOU2021060253、SHOU2021011001~SHOU2021011006、SHOU2021011011,体长72.51~110.53 mm;2021年6月采自浙江省杭州市余杭区径山镇(苕溪水系,119°48'52″E,30°25'33″N),2021年8月采自浙江省杭州市余杭区留下街道小和山(苕溪水系,120°0'31″E,30°12'46″N)。
形态描述: 测量标本10尾,相关可量、可数和可比性状见表 2。背鳍ⅲ-7~8,臀鳍ⅲ-9,胸鳍ⅰ-13~14,腹鳍ⅰ-7~8。侧线鳞8/41~44/3~4;背鳍前鳞16~18;围尾柄鳞12~15。下咽齿3行,2.3.5/5.3.2。
|
表 2 苕溪 |
体延长而侧扁,腹部圆,头长略大于体高。吻端稍钝圆。唇薄而光滑,无须。口端位,口裂接近眼中线下方,边缘较平直,向下倾斜。眼较大,位于头侧中上方,眼径与吻长近似相等,小于眼间距。背鳍无硬棘,位于体中部,外缘近平直。胸鳍末端尖,伸展不达腹鳍基部。腹鳍小,外缘呈弧形,成熟雄鱼可到达但不超过肛门,其起点与背鳍起点相对。臀鳍条前4根分枝鳍条延长,第2根最长,可超过尾柄末端。肛门紧靠臀鳍起点。侧线完全,在胸鳍上方向下弯曲,然后沿体侧下部逐渐向上延伸到尾柄中部末端。体侧鳞片中等大小,胸部与腹部鳞片细小,埋于皮下,不甚明显。
在繁殖季节,雄性在头部以及臀鳍会发育出发达的角质突起(追星),尤其在吻端至眼前下方及颊部追星最发达,会融合成条状。吻端至眼前7~8个大追星形成的融合条通常断裂成两段,颊部3~5颗大追星也会融合成一条。这2个追星条的顶端由各追星形成的棘突呈强锯齿状。下颌追星不发达,呈小颗粒状,每侧数颗排成1~2行。繁殖季节过后,追星脱落,留下圆形疤痕,一段时间后会完全消失。
体色: 成鱼生活时体色鲜艳,眼上缘黑色,头背部和下颌黑灰色。体背部灰黑色带橘黄色,腹部银白色。体侧具有由橙色条纹隔开的不规则蓝绿色横条纹4~8条。背鳍分枝鳍条分为三部分:基部鳍条和鳍间膜均黑色;中部鳍条透明,鳍间膜黑色;外缘鳍条黑色,鳍间膜透明。雄鱼偶鳍通常橘黄色,雌鱼偶鳍灰白色,鳍间膜夹杂许多细小黑斑点。臀鳍灰白色。尾鳍分叉,上下叶几乎等长,灰白色或浅黄色(图版-3,4)。
甲醛固定的样本头、背部灰黑色,腹部灰白色,背鳍黑色,尾鳍灰黑色(图版-1,2)。
地理分布:目前仅知分布于苕溪水系。
生境:生活于苕溪水系水流较平缓、底质为小型卵石或砾石的清澈浅水溪流中。
词源:新种种名“tiaoxiensis”为拉丁化的“苕溪的”意思,指该种的模式产地。
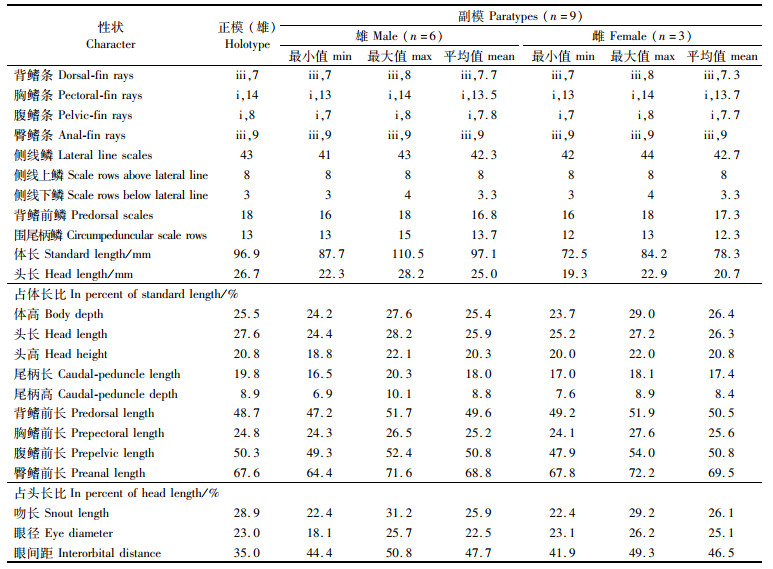
鉴别特征:新种与同一水系也有分布的棘颊






|
表 3 苕溪  |
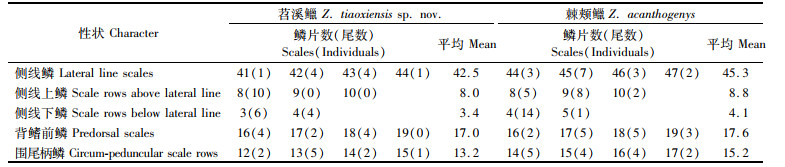
|
表 4 苕溪 |
全长1 140 bp的Cytb基因序列中无碱基的插入或缺失,其A、T、G、C的碱基含量平均值(不含外群)分别为26.7%、30.2%、16.0%、27.1%。A + T含量(56.9%)高于G + C含量(43. 1%),与鱼类线粒体基因A、T含量高,G、C含量低的特点一致。序列保守位点754个,占总位点数的66.1%;变异位点386个,占33.9%;单突变位点95个,占8.3%;简约信息位点291个,占25.5%。序列的转换颠换比为6.15。
以







|
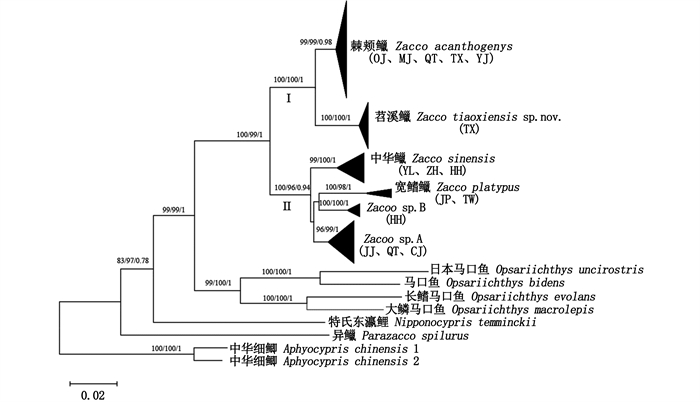
图 1 基于Cytb基因序列构建的马口鱼类邻接树, 节点上的数值对应NJ/ML/BI分析结果的支持率 Fig. 1 Phylogenetic relationships of Opsariichthine derived from NJ tree based on Cytb gene sequences, values at the nodes correspond to the support values for NJ/ML/BI methods |
基于双参数模型计算了



















基于Cytb基因全序列的系统发育关系分析进一步证实了以条纹特征分类的







综上所述,基于形态和遗传分析皆表明苕溪

| [1] |
陈宜瑜. 中国动物志, 硬骨鱼纲, 鲤形目(中)[M]. 北京: 科学出版社, 1998. CHEN Y Y. Fauna Sinica, Osteichthyes, Cypriniforms (Ⅱ)[M]. Beijing: Science Press, 1998. |
| [2] |
NELSON J S, GRANDE T C, WILSON M. Fishes of the world, 5th Edition[M]. Hoboken, New Jersey: John Wiley & Sons, 2016.
|
| [3] |
陈宜瑜. 马口鱼类分类的重新整理[J]. 海洋与湖沼, 1982(3): 293-299. CHEN Y Y. A revision of opsariichthine Cyprinid fishes[J]. Oceanologia et Limnologia Sinica, 1982(3): 293-299. |
| [4] |
杨慧兰, 王银肖, 谭慧敏, 等. 白洋淀流域宽鳍  遗传多样性及种群历史动态[J]. 上海海洋大学学报, 2021, 30(5): 837-846. 遗传多样性及种群历史动态[J]. 上海海洋大学学报, 2021, 30(5): 837-846.YANG H L, WANG Y X, TAN H M, et al. Genetic diversity and population historical dynamics of Zacco platypus in Baiyangdian Basin[J]. Journal of Shanghai Ocean University, 2021, 30(5): 837-846. |
| [5] |
BERREBI P, RETIF X, FANG F, et al. Population structure and systematics of Opsariichthys bidens (Osteichthyes: Cyprinidae) in south-east China using a new nuclear marker: the introns (EPIC-PCR)[J]. Biological Journal of the Linnean Society, 2005, 87(1): 155-166. |
| [6] |
PERDICES A, CUNHA C, COELHO M M. Phylogenetic structure of Zacco platypus (Teleostei, Cyprinidae) populations on the upper and middle Chang Jiang (=Yangtze) drainage inferred from cytochrome b sequences[J]. Molecular Phylogenetics and Evolution, 2004, 31(1): 192-203. DOI:10.1016/j.ympev.2003.07.001 |
| [7] |
PERDICES A, SAYANDA D, COELHO M M. Mitochondrial diversity of Opsariichthys bidens (Teleostei, Cyprinidae) in three Chinese drainages[J]. Molecular Phylogenetics and Evolution, 2005, 37(3): 920-927. DOI:10.1016/j.ympev.2005.04.020 |
| [8] |
LI G Y, WANG X Z, ZHAO Y H, et al. Speciation and phylogeography of Opsariichthys bidens (Pisces: Cypriniformes: Cyprinidae) in China: analysis of the cytochrome b gene of mtDNA from diverse populations[J]. Zoological Studies, 2009, 48(4): 569-583. |
| [9] |
PERDICES A, COELHO M M. Comparative phylogeography of Zacco platypus and Opsariichthys bidens (Teleostei, Cyprinidae) in China based on cytochrome b sequences[J]. Journal of Zoological Systematics and Evolutionary Research, 2006, 44: 330-338. DOI:10.1111/j.1439-0469.2006.00368.x |
| [10] |
CHEN I S, WU J H, HUANG S P. The taxonomy and phylogeny of the cyprinid genus Opsariichthys Bleeker (Teleostei: Cyprinidae) from Taiwan, with description of a new species[J]. Environmental Biology of Fishes, 2009, 86(1): 165-183. DOI:10.1007/s10641-009-9499-y |
| [11] |
HUANG S P, WANG F Y, WANG T Y. Molecular phylogeny of the Opsariichthys group (Teleostei: Cypriniformes) based on complete mitochondrial genomes[J]. Zoological Studies, 2017, 56(40): 1-13. |
| [12] |
BOULENGER G A. Descriptions of new freshwater fishes discovered by Mr. F. W. Stynn at Ningpo, China[J]. Proceedings of the Zoological Society of London, 1901, 70(2): 268-271. DOI:10.1111/j.1469-7998.1901.tb08544.x |
| [13] |
殷维. 马口鱼类的系统发育关系与生物地理研究[D]. 上海: 复旦大学, 2015. YIN W. Studies on phylogeny and biogeography of the opsariichthine fishes [D]. Shanghai: Fudan University, 2015. |
| [14] |
WANG X, LIU F, YU D, et al. Mitochondrial divergence suggests unexpected high species diversity in the opsariichthine fishes (Teleostei: Cyprinidae) and the revalidation of Opsariichthys macrolepis[J]. Ecology and Evolution, 2019, 9(5): 2664-2677. DOI:10.1002/ece3.4933 |
| [15] |
朱兰, 俞丹, 刘焕章. 中国北方  属鱼类一新种: 中华 属鱼类一新种: 中华 (鲤形目: 鲤科)[J]. 四川动物, 2020, 39(2): 168-176. (鲤形目: 鲤科)[J]. 四川动物, 2020, 39(2): 168-176.ZHU L, YU D, LIU H Z. Zacco sinensis sp. nov. (Cypriniformes: Cyprinidae), a new fish species from northern China[J]. Sichuan Journal of Zoology, 2020, 39(2): 168-176. |
| [16] |
陈义雄, 张咏青. 台湾淡水鱼类原色图鉴第一卷鲤形目[M]. 基隆: 水产出版社, 2005. CHEN Y X, ZHANG Y Q. A photographic guide to the Inland-water fishes of Taiwan Vol.1 Cypriniformes[M]. Keelung: The Sueichan Press, 2005. |
| [17] |
XIAO W H, ZHANG Y P, LIU H Z. Molecular systematics of Xenocyprinae (Teleostei: Cyprinidae) : taxonomy, biogeography and coevolution of a special group restricted in East Asia[J]. Molecular Phylogenetics and Evolution, 2001, 18(2): 163-173. |
| [18] |
SWINDELL S R, PLASTERER T N, SEQMAN. Contig assembly[J]. Methods in Molecular Biology, 1997, 70(70): 75. |
| [19] |
THOMPSON J D, GIBSON T J, PLEWNIAK F, et al. The Clustal X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 1997, 25(25): 4876-4882. |
| [20] |
TAMURA K, STECHER G, KUMAR S. MEGA11: Molecular evolutionary genetics analysis version 11.[J]. Molecular Biology and Evolution, 2021, 38(7): 7. |
| [21] |
KIMURA M. A simple method for estimation evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. Journal of Molecular Evolution, 1980, 16(2): 111-120. |
| [22] |
DARRIBA D, TABOADA G L, DOALLO R, et al. jModelTest 2: more models, new heuristics and parallel computing[J]. Nature Methods, 2012, 9(8): 772. |
| [23] |
STAMATAKIS A. RAxML Version 8: a tool for phylogenetic analysis and post-analysis of large phylogenies[J]. Bioinformatics, 2014, 30(9): 1312-1313. |
| [24] |
HUELSENBECK J P, RONQUIST F. MrBayes: Bayesian inference of phylogenetic trees[J]. Bioinformatics, 2001, 17(8): 754-755. |
| [25] |
HEE N Y, BYUNG S C, YANG S B. Geographic variation of body color and morphological characteristics of pale chub, Zacco platypus (Cyprinidae, Pisces)[J]. Korean Journal of Ichthyology, 2012, 24(3): 167-176. |
2. Engineering Research Center of Environmental DNA and Ecological Water Health Assessment, Shanghai Ocean University, Shanghai 201306, China;
3. Zhejiang Forest Resource Monitoring Center, Hangzhou 310020, Zhejiang, China;
4. Zhejiang Forestry Survey Planning and Design Company Limited, Hangzhou 310020, Zhejiang, China
 2023,
Vol. 32
2023,
Vol. 32



